背景介绍
本项目用于本地化查询、导出 HMDB 中的代谢物的信息。原始数据来自于:https://hmdb.ca/downloads (版本5.0,Data Set of All Metabolites),原始文件非常大,大约在 6G,先使用 Python 将原始代谢物的数据导入到 Mongo DB 中,然后再在Java项目中提供查询和导出。
解析代码
先要解决的一个问题就是如何读取一份超级大的XML文件,一次性读取到内存里显然不适合,虽然这次文件不大,只有6G,在生信分析的服务器动辄512G的内存前面来说就是弟弟,但必定开发还是在本地,而且保不齐后面文件还是会有更新
Python解析XML的库很多,用了 xml.etree.ElementTree 来处理,使用 iterparse 方法来迭代每一个标签,当遇到你需要处理的标签的时候,就来处理,并且使用了一个生成器(这里完全为了体验一下Python语法,也可以不用)
#!/usr/bin/python3
import xml.etree.ElementTree as ET
import psutil
import os
import xmltodict, json
from MongoDBUtil import MongoDBUtil
#这是原始xml里的namespace
namespace = '{http://www.hmdb.ca}';
def hmdbParse(filename, groupTag):
'''
增量解析 filename ,并且按groupTag作为一组,其实这里就是 metabolite 了
使用了生成器,并且使用
'''
ET.register_namespace("xmlns","http://www.hmdb.ca")
#迭代文件,增量
doc = ET.iterparse(filename, ('start', 'end'))
# Skip the root element
next(doc)
#循环读取解析器得到的结果
for event, elem in doc:
if event == 'end':
#一旦是结束标签,并且是你想要的标签,就return出去
if namespace+groupTag == elem.tag:
yield elem
#这里是把上一次的标签清空掉,这样就不会占用太多内存
elem.clear()
if __name__ == "__main__":
#xml文件地址,和需要解析的标签
data = hmdbParse('hmdb_l30000.xml', 'metabolite')
mongoUtil = MongoDBUtil(ip="127.0.0.1", db_name="bio_db", port="27017")
insertBatch = []
idx = 0
for meta in data:
#使用自带方法转换成xml字符串的字节流
xmlstr_byte = ET.tostring(meta, encoding='utf8', method='xml')
#转换成UTF8的xml字符
xmlstr = str( xmlstr_byte,encoding = "utf8" ).replace("xmlns:","");
#转成字典
xmldict = xmltodict.parse(xmlstr)
accessionId = meta.findtext(namespace+"accession")
#批量插入
insertBatch.append(xmldict)
idx+=1
if(idx % 500 == 0):
#500条插入一次
mongoUtil.insert_many(collect_name="hmdb2210", documents=insertBatch)
#插入后清理一下
insertBatch.clear()
print("insert 500 data, clear")
#如果循环执行完了,insertbatch中还有小于500的,那就执行完,因为我也不知道
#怎么判断生成器是不是执行到了最后一个行
if(len(insertBatch) < 500):
mongoUtil.insert_many(collect_name="hmdb2210", documents=insertBatch)
print('all data done.')
mongodb的写入问题
在第一个版本中,我是每一条解析出来的metabolite都会插入的mongo中去,结果执行了将近5个小时,才导入7万多条数据(最后实测下来是23万条左右的数据),于是就停掉了,来找原因。
db.hmdb2210.find().count()
229920
找到一篇文章,说的非常棒,https://www.cnblogs.com/wodeboke-y/p/10828985.html 这个是我看到的地址,但这也是转载的,文章里的原文链接已经无法访问了。
大概是2个意思:
- 大批量插入的时候,不建议用自己的ID
- 大批量插入的时候,建议使用批量插入功能,而不是单条插入的功能
当在不指定_id插入数据的时候,其_id是系统自动计算生成的。MongoDB通过计算机特征值、时间、进程ID与随机数来确保生成的_id是唯一的。而在指定_id插入时,MongoDB每插一条数据,都需要检查此_id可不可用,当数据库中数据条数太多的时候,这一步的查询开销会拖慢整个数据库的插入速度。
最后我放弃了使用数据ID的想法,也把单条插入改成了批量插入,大约500条一个批次,最后导入的时间大概在40分钟。(7万条5小时,22万条40分钟)
读取和导出
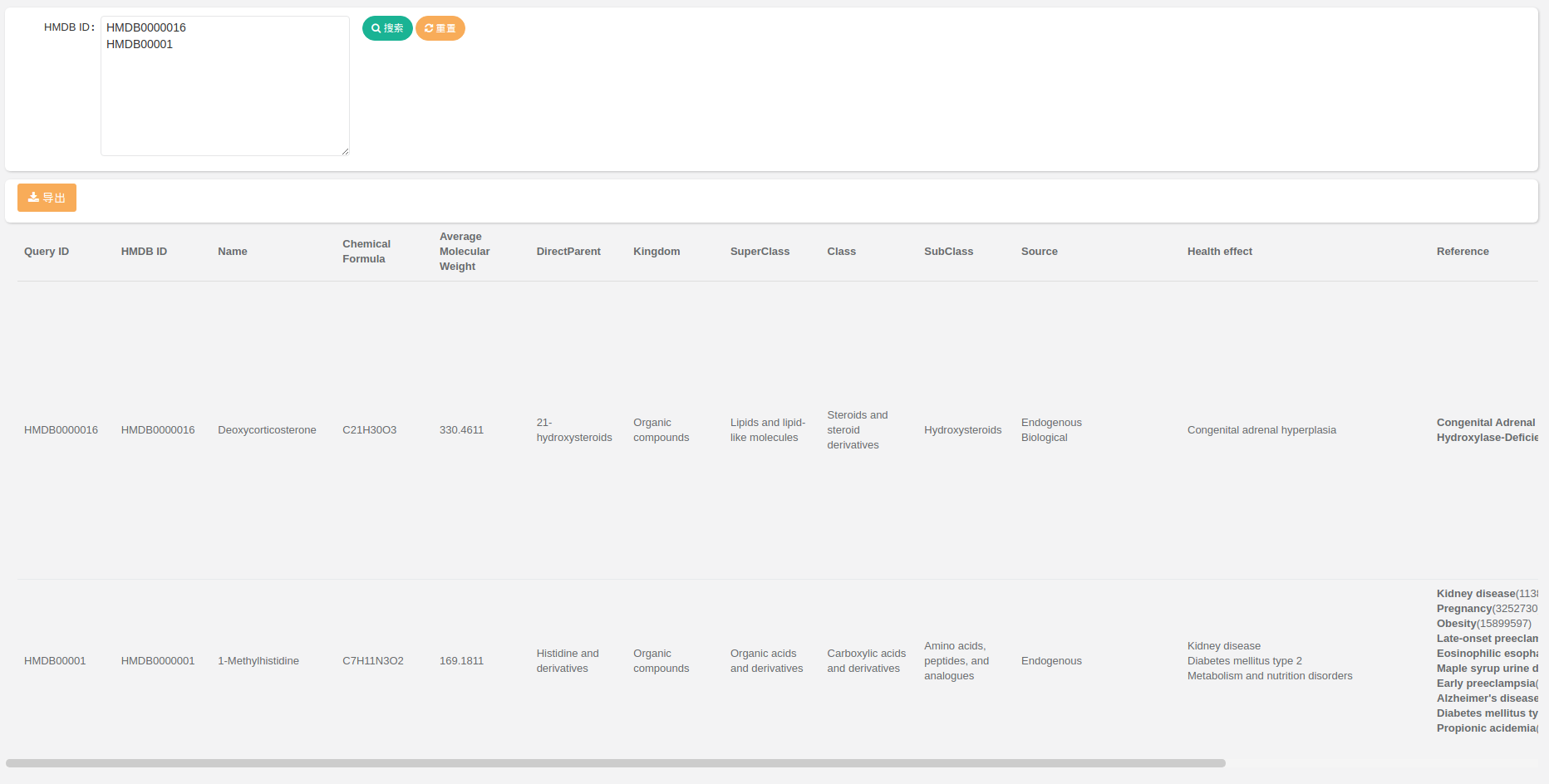
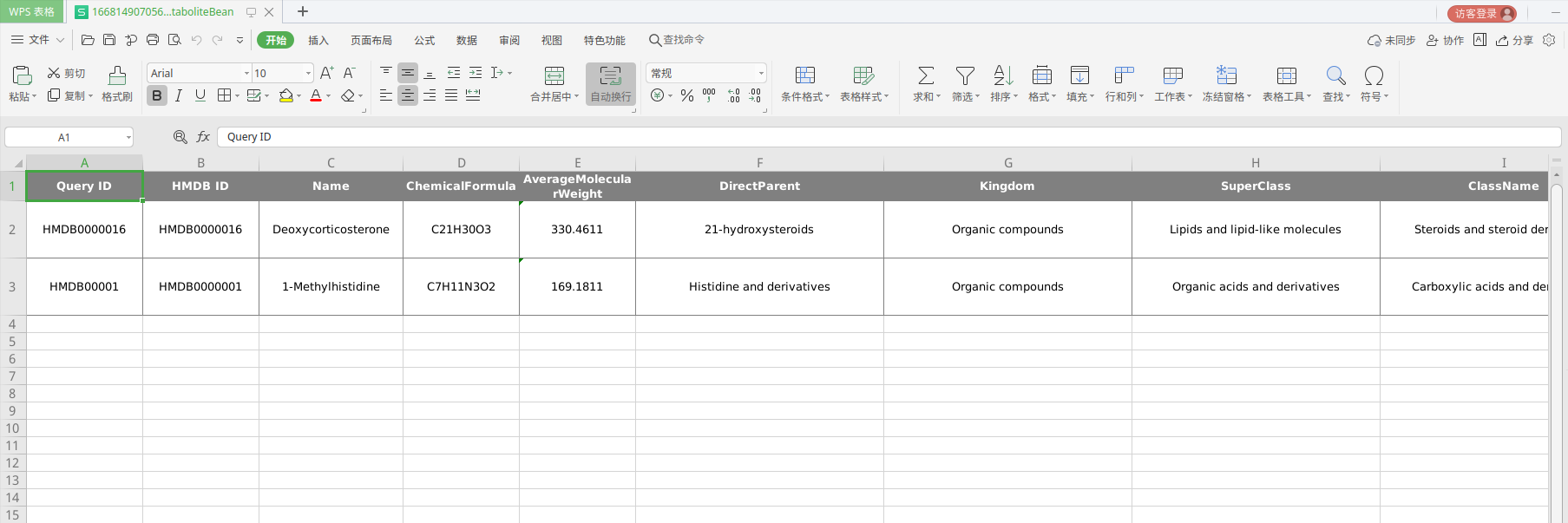
读取和导出是建立在RuoYi项目上的,因为手边有这个脚手架,开发起来很方便,主要解析了几个数据字段并导出
- Chemical Formula
- AverageMolecularWeight
- DirectParent
- Kingdom
- SuperClass
- Class
- SubClass
- Source
- Health effect
- Reference
- Pathway
已经将代码开源到 github https://github.com/5venw0ng/hmdblocal_tools.git
效果预览